Prot2Chat:融合蛋白质序列与结构的新型问答框架
蛋白质在生物体中起着关键作用,但理解其功能面临诸多挑战,包括分类方法的灵活性不足、无法有效利用空间结构信息,以及缺乏系统的蛋白质问答系统评估指标。为了解决这些问题,本文提出了Prot2Chat,这是一
论文标题:
Prot2Chat: Protein LLM with Early Fusion of Sequence and Structure
论文链接:
https://arxiv.org/abs/2502.06846
模型链接:
https://www.modelscope.cn/models/wangzc025/prot2chat/files
01.论文解读
蛋白质在生物体中起着关键作用,但理解其功能面临诸多挑战,包括分类方法的灵活性不足、无法有效利用空间结构信息,以及缺乏系统的蛋白质问答系统评估指标。为了解决这些问题,本文提出了Prot2Chat,这是一个将蛋白质序列和结构信息与自然语言结合的新型框架,通过统一模块实现多模态蛋白质表示与自然语言的融合,并利用大语言模型(LLM)驱动答案生成。采用改进的ProteinMPNN编码器,以统一方式编码蛋白质序列和结构信息,并通过蛋白质-文本适配器和LLaMA3解码器实现跨注意力机制。为优化训练效率,本文作者冻结了编码器,并在解码器中应用了LoRA技术。实验结果表明,该模型在两个数据集上均表现出色,零样本预测结果也展示了其强大的泛化能力。
02.主要方法
2.1 序列和结构融合的蛋白质编码器作者改进了ProteinMPNN编码器,实现了序列和结构的早期融合。ProteinMPNN原本用于基于骨架结构设计蛋白质序列,输入为蛋白质残基原子的3D坐标。作者通过在解码器中引入序列嵌入,使得序列和结构信息在模型初始化时即融合。最终的蛋白质节点向量作为蛋白质特征输入到适配器中。
2.2 蛋白质-文本适配器
适配器通过线性投影层、位置编码和跨注意力机制将蛋白质编码器的信息与自然语言对齐。通过引入可学习的查询向量,适配器能够提取蛋白质特征中的关键语义信息,并将其转换为蛋白质提示。随后将蛋白质提示与文本问题结合后输入到LLM中,通过LoRA技术对LLM进行微调,以提高模型的领域适应性。Prot2Chat的总训练参数为93M,显著低于其他现有模型。
03.主要改进
3.1 改进现有的结构编码器 ProteinMPNN,无需训练即可实现结构和序列的早期融合。
3.2 基于这个蛋白质编码器,我们实现了一个仅含 93M 个训练参数的轻量级高效蛋白质大语言模型,能够实现蛋白质和文本信息的早期融合。
3.3 我们在各种评估数据集上进行了系统评估,以验证模型的生成能力和泛化能力。评估方式包括传统指标评估、在线大模型评估和人工专家评估。
04.解决的问题
该方法主要为解决蛋白质问答系统中分类方法生成内容灵活性不足、无法有效利用空间结构信息,以及缺乏系统的蛋白质问答系统评估指标等问题提出了新实现路径。
05.论文实验
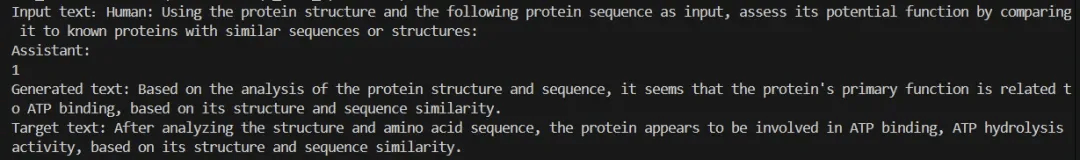
本文通过利用Mol-Instructions和UniProtQA数据集以及多种不同的基线模型(如LLaMA3和BioMedGPT)来进行对比试验;并且使用BLEU和ROUGE等传统指标评估文本生成效果的同时利用在线KIMI模型评估生成答案与目标文本的相似度。还进行了专家手动评估,以验证模型输出的质量。
实验结果表明,直接将蛋白质序列输入LLM会导致生成的文本混乱无序。而整合结构信息后,模型性能显著提升。Prot2Chat在Mol-Instructions和UniProtQA数据集上的表现均优于其他基线模型,验证了其强大的泛化能力。
06.模型实践
- Download Prot2Chat repository from https://www.modelscope.cn/models/wangzc025/prot2chat/files
- Prepare environment for Prot2Chat in prot2chat/requirement.sh
- Run generation demo
python generate.py
-base_model_path=your_path_to_base_model
-lora_weight_path=your_path_to_lora_weight.pth
-pdb_path=your_path_to_pdbs
-checkpoint_path=your_path_to_adapter_checkpoint
-conversation_data_path=your_path_to_QAdata_test.json
点击链接, 即可跳转模型链接~
https://www.modelscope.cn/models/wangzc025/prot2chat/files
更多推荐
 已为社区贡献634条内容
已为社区贡献634条内容










所有评论(0)